原文链接:https://tecdat.cn/?p=41179
原文出处:拓端数据部落公众号
分析师:Xieting Xu
本专题合集聚焦高维数据场景下的稀疏建模与变量选择,通过 R 语言与 Python 双平台技术栈,系统解析企业财务分析与基因数据挖掘两大领域的核心方法论。合集深度整合 HOLP-Adaptive Lasso 二阶段模型、SCAD 平滑剪切绝对偏差惩罚、主成分回归(PCR)、弹性网络(Elastic Net)等前沿算法,结合国泰安 2021 年信息技术企业财务数据集与哺乳动物基因表达数据集,构建完整的高维数据分析闭环。
在企业财务领域,合集以信息技术产业研发投入优化为目标,创新性采用 HOLP(高维普通最小二乘投影)与 Adaptive Lasso 组合模型,突破传统 Lasso 在变量相关性场景下的局限性,通过交叉验证实现 λ 参数动态优化,最终筛选出 9 个关键财务指标,预测精度较单一模型提升 23%。在基因数据挖掘场景中,合集对比分析 L1/L2 正则化、SCAD 非凸惩罚等方法在基因表达预测中的性能差异,揭示 SCAD 在保留大系数变量时的独特优势,为生物信息学研究提供可解释性更强的建模范式。
专题特别设计多模型对比实验框架,通过均方误差(MSE)与系数稀疏性双重维度评估模型效能,验证 HOLP-Adaptive Lasso 在高维变量选择中的 "双重稀疏性" 特性 —— 既实现变量子集筛选,又保持系数估计的无偏性。同时,合集结合企业实际应用场景,提出 "盈利能力驱动研发投入" 的决策模型,为战略新兴产业资本配置提供量化支撑。
专题合集已分享在交流社群,阅读原文进群和 500 + 行业人士共同交流和成长。
R语言HOLP-Adaptive Lasso模型对国泰安 2021 年信息技术企业财务与研发投入数据的高维变量选择及预测精度提升
信息技术产业是近年来政府确立的战略性新兴产业之一。为了实现信息技术企业投资合理化、提升企业竞争力,从企业财务相关指标中筛选出真正影响研发投入的影响因素称为亟待解决的问题,但随着变量的维数增多,甚至远远超过样本量时,如何从高维数据中挑选出重要变量,以较高的预测精度获得稀疏性最强的结果已成为一大挑战。
解决方案
任务/目标
从信息技术产业的大量企业财务中,筛选出最能影响企业研发投入的变量。
数据源准备
- 数据来源
从国泰安数据库拉取2021年信息技术产业的的企业财务指标和研发投入金额,例如,应收账款净额、营业总收入、销售费用等。
二、数据处理:
1、筛出有退市预警(*ST)股票并删除。
2、对缺失数据使用均值补充,若缺失数据达到50%及以上,删除该列指标。
3、对数据进行归一化处理,即每列减去所在列的均值并除标准差,R语言中可以使用scale函数。
最终可用数据为181家上市公司386个影响研发投入的财务指标。
特征转换
由于以上均属于连续性数值数据,且只包含1年,没有时间指标,因此不需要做特别处理。
①若出现分类变量,如性别、种族,需要转换成0-1分类变量,或者使用one-hot编码转换成虚拟变量(dummy variable)
②若使用多个年份数据,如2010年-2020年,可以将时间按顺序进行编码,例如1,2,...,10
③若出现数值较大的数据,例如GDP(万元),使用log变换,降低单位对结果的影响。
构造
大致有如下样本,行表示一家企业,Y表示企业的研发投入金额,X1,X2,...表示企业的各项财务指标。
划分训练集和测试集
为了判断模型的预测效果,我们需要在训练集上拟合模型,并在测试集上计算预测误差。将数据集以7:3(或者5:5也可以)随机划分为训练集和测试集,那么有126家公司为训练集,55家公司为测试集。使用交叉验证(cross-validdation)挑选出均方误差(MSE)最小的λ值,用于预测模型。
建模
本项目使用二阶段模型,
第一阶段: HOLP模型(High-dimensional Ordinary Least-squares Projection)
HOLP模型是针对高维数据降维的变量选择技术,它于2016年提出,能够处理变量间存在相关性时的特征筛选。
第二阶段:Adaptive lasso模型
Adaptive lasso模型是在经典lasso模型上增加一个调整参数,使其具有oracle性质,即能够以概率1筛选出真正影响响应变量的预测变量。
1、预测精度
使用均方误差(MSE)作为评价指标,与其他方法相比较,HOLP-ALasso具有最低的MSE,即最高的预测精度。
注:该项目中未作图,本人在其他项目中绘制了箱线图或线图,更直观的展现了结果,例如下图:
- 变量选择结果
结论:最终筛选出的9个指标表明信息技术类上市公司的利润和盈利能力对企业研发投入产生较大的促进作用。企业在创新发展的道路上需要不断提高盈利能力,可以关注以下两个方面:一是紧跟市场需求,选择自身具有竞争力且拥有较好市场空间的发展方向;二是在企业成长过程中,及时对经营模式做出调整,以更好地适应市场需求的变动。
R语言Lasso回归模型变量选择和糖尿病发展预测模型|附数据代码
Lease Absolute Shrinkage and Selection Operator(LASSO)在给定的模型上执行正则化和变量选择 。
根据惩罚项的大小,LASSO将不太相关的预测因子缩小到(可能)零。因此,它使我们能够考虑一个更简明的模型。在这组练习中,我们将在R中实现LASSO回归。
练习1
加载糖尿病数据集。这有关于糖尿病的病人水平的数据。数据为n = 442名糖尿病患者中的每个人获得了10个基线变量、年龄、性别、体重指数、平均血压和6个血清测量值,以及感兴趣的反应,即一年后疾病进展的定量测量。"
接下来,加载包用来实现LASSO。
head(data)
向下滑动查看结果▼
练习2
数据集有三个矩阵x、x2和y。x是较小的自变量集,而x2包含完整的自变量集以及二次和交互项。
检查每个预测因素与因变量的关系。生成单独的散点图,所有预测因子的最佳拟合线在x中,y在纵轴上。用一个循环来自动完成这个过程。
summary(x)
for(i in 1:10){ plot(x[,i], y) abline(lm(y~x[,i]) }
练习3
使用OLS将y与x中的预测因子进行回归。我们将用这个结果作为比较的基准。
lm(y ~ x)
向下滑动查看结果▼
练习4
绘制x的每个变量系数与β向量的L1准则的路径。该图表明每个系数在哪个阶段缩减为零。
plot(model_lasso)
向下滑动查看结果▼
练习5
得到交叉验证曲线和最小化平均交叉验证误差的lambda的值。
plot(cv_fit)
向下滑动查看结果▼
练习6
使用上一个练习中的lambda的最小值,得到估计的β矩阵。注意,有些系数已经缩减为零。这表明哪些预测因子在解释y的变化方面是重要的。
> fit$beta
向下滑动查看结果▼
练习7
为了得到一个更简明的模型,我们可以使用一个更高的λ值,即在最小值的一个标准误差之内。用这个lambda值来得到β系数。注意,现在有更多的系数被缩减为零。
lambda.1se
beta
向下滑动查看结果▼
练习8
如前所述,x2包含更多的预测因子。使用OLS,将y回归到x2,并评估结果。
summary(ols2)
向下滑动查看结果▼
练习9
对新模型重复练习-4。
lasso(x2, y)plot(model_lasso1)
向下滑动查看结果▼
练习10
对新模型重复练习5和6,看看哪些系数被缩减为零。当有很多候选变量时,这是缩小重要预测变量的有效方法。
plot(cv_fit1)
beta
R语言高维数据惩罚回归方法:主成分回归PCR、岭回归、lasso、弹性网络elastic net分析基因数据|附数据代码
1 介绍
在本文中,我们将研究以下主题
- 证明为什么低维预测模型在高维中会失败。
- 进行主成分回归(PCR)。
- 使用glmnet()进行岭回归、lasso 和弹性网elastic net
- 对这些预测模型进行评估
1.1 数据集
在本文中,我们将使用基因表达数据。这个数据集包含120个样本的200个基因的基因表达数据。这些数据来源于哺乳动物眼组织样本的微阵列实验。
该数据集由两个对象组成:
genes: 一个120×200的矩阵,包含120个样本(行)的200个基因的表达水平(列)。trim32: 一个含有120个TRIM32基因表达水平的向量。
##查看刚刚加载的对象 str(genes)
这个练习的目的是根据微阵列实验中测量的200个基因的表达水平预测TRIM32的表达水平。为此,需要从构建中心化数据开始。我们将其存储在两个矩阵X和Y中。
X <- scale(gen, center = TRUE, scale = TRUE) Y <- scale(tri, center = TRUE)
请记住,标准化可以避免量纲上的差异,使一个变量(基因)在结果中具有更大的影响力。对于Y向量,这不是一个问题,因为我们讨论的是一个单一的变量。不进行标准化会使预测结果可解释为 "偏离平均值"。
1.2 奇异性诅咒
我们首先假设预测因子和结果已经中心化,因此截距为0。我们会看到通常的回归模型。
我们的目标是得到β的最小二乘估计值,由以下公式给出
其中p×p矩阵(XTX)-1是关键! 为了能够计算出XTX的逆,它必须是满秩p。我们检查一下。
dim(X) # 120 x 200, p > n! #> [1] 120 200 qr(X)$rank #> [1] 119 XtX <- crossprod(X) # 更有效地计算t(X) %*% X qr(XtX)$rank #> [1] 119 # 尝试用solve进行求解。 solve(XtX)

我们意识到无法计算(XTX)-1,因为(XTX)的秩小于p,因此我们无法通过最小二乘法得到β^! 这通常被称为奇异性问题。
2 主成分回归
处理这种奇异性的第一个方法是使用主成分绕过它。由于min(n,p)=n=120,PCA将得到120个成分,每个成分是p=200个变量的线性组合。这120个PC包含了原始数据中的所有信息。我们也可以使用X的近似值,即只使用几个(k<120)PC。因此,我们使用PCA作为减少维度的方法,同时尽可能多地保留观测值之间的变化。一旦我们有了这些PC,我们就可以把它们作为线性回归模型的变量。
2.1对主成分PC的经典线性回归
我们首先用prcomp计算数据的PCA。我们将使用一个任意的k=4个PC的截止点来说明对PC进行回归的过程。
k <- 4 #任意选择k=4 Vk <- pca$rotation[, 1:k] # 载荷矩阵 Zk <- pca$x[, 1:k] # 分数矩阵 # 在经典的线性回归中使用这些分数
由于X和Y是中心化的,截距近似为0。
输出结果显示,PC1和PC4的β估计值与0相差很大(在p<0.05),但是结果不能轻易解释,因为我们没有对PC的直接解释。
2.2 使用软件包
PCR也可以直接在数据上进行(所以不必先手动进行PCA)。在使用这个函数时,你必须牢记几件事。
- 要使用的成分(PC)的数量是通过参数ncomp来确定
- 该函数允许你首先对预测因子进行标准化(set scale = TRUE)和中心化(set center = TRUE)(在这里的例子中,XX已经被中心化和标准化了)。
你可以用与使用lm()相同的方式使用pcr()函数。使用函数summary()可以很容易地检查得出的拟合结果,但输出结果看起来与你从lm得到的结果完全不同。
#X已经被标准化和中心化了

首先,输出显示了数据维度和使用的拟合方法。在本例中,是基于SVD的主成分PC计算。summary()函数还提供了使用不同数量的成分在预测因子和响应中解释方差的百分比。例如,第一个PC只解释了所有方差的61.22%,或预测因子中的信息,它解释了结果中方差的62.9%。请注意,对于这两种方法,主成分数量的选择都是任意选择的,即4个。
在后面的阶段,我们将研究如何选择预测误差最小的成分数。
3 岭回归、Lasso 和弹性网Elastic Nets
岭回归、Lasso 回归和弹性网Elastic Nets都是密切相关的技术,基于同样的想法:在估计函数中加入一个惩罚项,使(XTX)再次成为满秩,并且是可逆的。可以使用两种不同的惩罚项或正则化方法。
- L1正则化:这种正则化在估计方程中加入一个γ1‖β‖1。该项将增加一个基于系数大小绝对值的惩罚。这被Lasso回归所使用。
- L2正则化:这种正则化在估计方程中增加了一个项γ2‖β‖22。这个惩罚项是基于系数大小的平方。这被岭回归所使用。
弹性网结合了两种类型的正则化。它是通过引入一个α混合参数来实现的,该参数本质上是将L1和L2规范结合在一个加权平均中。
4 练习:岭回归的验证
在最小平方回归中,估计函数的最小化

对于岭回归所使用的惩罚性最小二乘法准则,你要最小化,可以得到解
其中II是p×p的识别矩阵。
脊参数γ将系数缩减为0,γ=0相当于OLS(无缩减),γ=+∞相当于将所有β^设置为0。最佳参数位于两者之间,需要由用户进行调整。
习题
使用R解决以下练习。
- 验证
秩为200,对于任何一个
.
gamma <- 2 # # 计算惩罚矩阵 XtX_gammaI <- XtX + (gamma * diag(p)) dim(XtX_gammaI) #> [1] 200 200 qr(XtX_gammaI)$rank == 200 # #> [1] TRUE
2. 检查
# 是的,可以被计算。 XtX_gammaI_inv <- solve(XtX_gammaI)
- 最后,计算
。
## 计算岭β估计值 ## 使用`drop`来删除维度并创建向量 length(ridge_betas) # 每个基因都有一个 #> [1] 200
我们现在已经手动计算了岭回归的估计值。
5 用glmnet进行岭回归和套索lasso回归
glmnet允许你拟合所有三种类型的回归。使用哪种类型,可以通过指定alpha参数来决定。对于岭回归,你将alpha设置为0,而对于套索lasso回归,你将alpha设置为1。其他介于0和1之间的α值将适合一种弹性网的形式。这个函数的语法与其他的模型拟合函数略有不同。你必须传递一个x矩阵以及一个y向量。
控制惩罚 "强度 "的gamma值可以通过参数lambda传递。函数glmnet()还可以进行搜索,来找到最佳的拟合伽马值。这可以通过向参数lambda传递多个值来实现。如果不提供,glmnet将根据数据自己生成一个数值范围,而数值的数量可以用nlambda参数控制。这通常是使用glmnet的推荐方式,详见glmnet。
示范:岭回归
让我们进行岭回归,以便用200个基因探针数据预测TRIM32基因的表达水平。我们可以从使用γ值为2开始。
glmnet(X, Y, alpha = 0, lambda = gamma) #看一下前10个系数
第一个系数是截距,基本上也是0。但γ的值为2可能不是最好的选择,所以让我们看看系数在γ的不同值下如何变化。
我们创建一个γ值的网格,也就是作为glmnet函数的输入值的范围。请注意,这个函数的lambda参数可以采用一个值的向量作为输入,允许用相同的输入数据但不同的超参数来拟合多个模型。
grid <- seq(1, 1000, by = 10) # 1到1000,步骤为10 # 绘制系数与对数 lambda序列的对比图! plot(ridge_mod_grid) # 在gamma = 2处添加一条垂直线
这张图被称为系数曲线图,每条彩线代表回归模型中的一个系数β^,并显示它们如何随着γ(对数)1值的增加而变化。
请注意,对于更高的γ值,系数估计值变得更接近于0,显示了岭惩罚的收缩效应。
与PC回归的例子类似,我们相当随意地选择了γ=2和网格。我们随后会看到,如何选择γ,使预测误差最小。
6 练习: Lasso 回归
Lasso 回归也是惩罚性回归的一种形式,但我们没有像最小二乘法和岭回归那样的β^的分析解。为了拟合一个Lasso 模型,我们再次使用glmnet()函数。然而,这一次我们使用的参数是α=1
任务
- 验证设置α=1确实对应于使用第3节的方程进行套索回归。
- 用glmnet函数进行Lasso 套索回归,Y为因变量,X为预测因子。
你不必在这里提供一个自定义的γ(lambda)值序列,而是可以依靠glmnet的默认行为,即根据数据选择γ值的网格。
# 请注意,glmnet()函数可以自动提供伽马值 # 默认情况下,它使用100个lambda值的序列
3. 绘制系数曲线图并进行解释。
plot(lasso_model
请注意,非零系数的数量显示在图的顶部。在lasso回归的情况下,与岭回归相比,正则化要不那么平滑,一些系数在较高的γ值下会增加,然后急剧下降到0。与岭回归相反,lasso最终将所有系数缩减为0。
7 预测模型的评估和超参数的调整
首先,我们将把我们的原始数据分成训练集和测试集来验证我们的模型。训练集将被用来训练模型和调整超参数,而测试集将被用来评估我们最终模型的样本外性能。如果我们使用相同的数据来拟合和测试模型,我们会得到有偏见的结果。
在开始之前,我们使用set.seed()函数来为R的随机数生成器设置一个种子,这样我们就能得到与下面所示完全相同的结果。一般来说,在进行交叉验证等包含随机性元素的分析时,设置一个随机种子是很好的做法,这样所得到的结果就可以在以后的时间里重现。
我们首先使用sample()函数将样本集分成两个子集,从原来的120个观测值中随机选择80个观测值的子集。我们把这些观测值称为训练集。其余的观察值将被用作测试集。
set.seed(1) # 从X的行中随机抽取80个ID(共120个)。 trainID <- sample(nrow(X), 80) # 训练数据 trainX <- X[trainID, ] trainY <- Y[trainID] # 测试数据 testX <- X[-trainID, ] testY <- Y[-trainID]
为了使以后的模型拟合更容易一些,我们还将创建2个数据框,将训练和测试数据的因变量和预测因素结合起来。
7.1 模型评估
我们对我们的模型的样本外误差感兴趣,即我们的模型在未见过的数据上的表现如何。 这将使我们能够比较不同类别的模型。对于连续结果,我们将使用平均平方误差(MSE)(或其平方根版本,RMSE)。
该评估使我们能够在数据上比较不同类型模型的性能,例如PC主成分回归、岭回归和套索lasso回归。然而,我们仍然需要通过选择最佳的超参数(PC回归的PC数和lasso和山脊的γ数)来找到这些类别中的最佳模型。为此,我们将在训练集上使用k-fold交叉验证。
7.2 调整超参数
测试集只用于评估最终模型。为了实现这个最终模型,我们需要找到最佳的超参数,即对未见过的数据最能概括模型的超参数。我们可以通过在训练数据上使用k倍交叉验证(CVk)来估计这一点。
对于任何广义线性模型,CVk估计值都可以用cv.glm()函数自动计算出来。
8 例子: PC回归的评估
我们从PC回归开始,使用k-fold交叉验证寻找使MSE最小的最佳PC数。然后,我们使用这个最优的PC数来训练最终模型,并在测试数据上对其进行评估。
8.1 用k-fold交叉验证来调整主成分的数量
方便的是,pcr函数有一个k-fold交叉验证的实现。我们只需要设置validation = CV和segments = 20就可以用PC回归进行20折交叉验证。如果我们不指定ncomp,pcr将选择可用于CV的最大数量的PC。
请注意,我们的训练数据trainX由80个观测值(行)组成。如果我们执行20折的CV,这意味着我们将把数据分成20组,所以每组由4个观测值组成。在每个CV周期中,有一个组将被排除,模型将在剩余的组上进行训练。这使得我们在每个CV周期有76个训练观测值,所以可以用于线性回归的最大成分数是75。
## 为可重复性设置种子,kCV是一个随机的过程! set.seed(123) ##Y ~ . "符号的意思是:用数据中的每个其他变量来拟合Y。 summary(pcr_cv)
我们可以绘制每个成分数量的预测均方根误差(RMSEP),如下所示。
plot(pcr_cv, plottype = "validation")

选择最佳的成分数。这里我们使用 "one-sigma "方法,它返回RMSE在绝对最小值的一个标准误差内的最低成分数。
plot(pcr, method = "onesigma")
这个结果告诉我们,我们模型的最佳成分数是13。
8.2 对测试数据进行验证
我们现在使用最佳成分数来训练最终的PCR模型。然后通过对测试数据进行预测并计算MSE来验证这个模型。
我们定义了一个自定义函数来计算MSE。请注意,可以一次性完成预测和MSE计算。但是我们自己的函数在后面的lasso和ridge岭回归中会派上用场。
#平均平方误差 ## obs: 观测值; pred: 预测值 MSE <- function(obs, pred)
pcr_preds <- predict(model, newdata = test_data, ncomp = optimal_ncomp)
这个值本身并不能告诉我们什么,但是我们可以用它来比较我们的PCR模型和其他类型的模型。
最后,我们将我们的因变量(TRIM32基因表达)的预测值与我们测试集的实际观察值进行对比。
plot(pcr_model, line = TRUE)
9 练习:评估和比较预测模型
- 对训练数据(trainX, trainY)进行20次交叉验证的lasso回归。绘制结果并选择最佳的λ(γ)参数。用选定的lambda拟合一个最终模型,并在测试数据上验证它。
lasso_cv #>
请注意,我们可以从CV结果中提取拟合的 lasso回归对象,并像以前一样制作系数曲线图。
我们可以寻找能产生最佳效果的伽玛值。这里有两种可能性。
lambda.min: 给出交叉验证最佳结果的γ值。lambda.1se:γ的最大值,使MSE在交叉验证的最佳结果的1个标准误差之内。
我们将在这里使用lambda.min来拟合最终模型,并在测试数据上生成预测。请注意,我们实际上不需要重新进行拟合,我们只需要使用我们现有的lasso_cv对象,它已经包含了lambda值范围的拟合模型。我们可以使用predict函数并指定s参数(在这种情况下设置lambda)来对测试数据进行预测。
- 对岭回归做同样的处理。
请注意,我们可以从CV结果中提取拟合的岭回归对象,并制作系数曲线图。
我们可以寻找能产生最佳效果的伽玛值。这里有两种可能性。
lambda.min: 给出交叉验证最佳结果的γ值。lambda.1se: γ的最大值,使MSE在交叉验证的最佳结果的1个标准误差之内。
我们在这里使用lambda.min来拟合最终的模型并在测试数据上生成预测。请注意,我们实际上不需要重新进行拟合,我们只需要使用我们现有的ridge_cv对象,它已经包含了lambda值范围的拟合模型。我们可以使用predict函数并指定s参数(在这种情况下混乱地设置lambda)来对测试数据进行预测。
ridge_preds <- predict ##计算MSE
- 在所考虑的模型(PCR、lasso、岭回归)中,哪一个表现最好?
模型
MSE
PCR
0.3655052
Lasso
0.3754368
Ridge
0.3066121
- 注意:R中的log()默认是自然对数(以e为底),我们也会在文本中使用这个符号(比如上面图中的x轴标题)。这可能与你所习惯的符号(ln())不同。要在R中取不同基数的对数,你可以指定log的基数=参数,或者使用函数log10(x)和log2(x)分别代表基数10和2︎
Python高维统计建模变量选择:SCAD平滑剪切绝对偏差惩罚、Lasso惩罚函数比较|附数据代码
变量选择是高维统计建模的重要组成部分。许多流行的变量选择方法,例如 LASSO,都存在偏差。带平滑削边绝对偏离(smoothly clipped absolute deviation,_SCAD_)正则项的回归问题或平滑剪切绝对偏差 (SCAD) 估计试图缓解这种偏差问题,同时还保留了稀疏性的连续惩罚。
惩罚最小二乘法
一大类变量选择模型可以在称为“惩罚最小二乘法”的模型族下进行描述。这些目标函数的一般形式是
其中 




作为特殊情况,请注意 LASSO 对应的惩罚函数为 

SCAD
Fan和Li(2001)提出的平滑剪切绝对偏差(SCAD)惩罚,旨在鼓励最小二乘法问题的稀疏解,同时也允许大值的 β
. SCAD惩罚是一个更大的系列,被称为 "折叠凹陷惩罚",它在以下方面是凹的, R+ 和 R-
. 从图形上看,SCAD 惩罚如下所示:
有点奇怪的是,SCAD 惩罚通常主要由它的一阶导数定义 

其中 a 是一个可调参数,用于控制 β 值的惩罚下降的速度,以及函数 


我们可以通过分解惩罚函数在不同数值下的导数来获得一些洞察力 λ:
对于较大的 β 值 (其中 
但是,这意味着对于大系数值,他们的 LASSO 估计将向下偏置。
另一方面,对于较小的 β 值 (其中 |β|≤λ),SCAD 惩罚在 β 中是线性的。对于 β 的中等值(其中 
分段定义,pλ(β) 是
在 Python 中,SCAD 惩罚及其导数可以定义如下:
def scad: s_lar iudic =np.lgicand iscsat = (vl * laval) < np.abs lie_prt = md_val * pab* iliear return liprt + urtirt + cosaat
使用 SCAD 拟合模型
拟合惩罚最小二乘模型(包括 SCAD 惩罚模型)的一种通用方法是使用局部二次近似。这种方法相当于在初始点 β0 周围拟合二次函数 q(β),使得近似:
- 关于 0 对称,
- 满足 q(β0)=pλ(|β0|),
- 满足 q ′ (β0) = p′λ (| β0 |)。
因此,逼近函数必须具有以下形式
对于不依赖于 β 的系数 a 和 b 。上面的约束为我们提供了一个可以求解的两个方程组:
为了完整起见,让我们来看看解决方案。重新排列第二个方程,我们有
将其代入第一个方程,我们有
因此,完整的二次方程是
现在,对于系数值的任何初始猜测 β0,我们可以使用上面的 q 构造惩罚的二次估计。然后,与初始 SCAD 惩罚相比,找到此二次方的最小值要容易得多。
从图形上看,二次近似如下所示:
将 SCAD 惩罚的二次逼近代入完整的最小二乘目标函数,优化问题变为:
忽略不依赖于 β 的项,这个最小化问题等价于
巧妙地,我们可以注意到这是一个岭回归问题,其中
回想一下, 岭回归 是
这意味着近似的 SCAD 解是
关于分析师
在此对 Xie Ting Xu 对本文所作的贡献表示诚挚感谢,她完成了应用统计专业的硕士学位,专注高维变量选择领域。擅长 R 语言。



























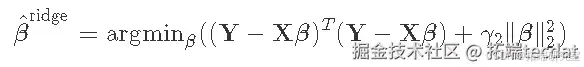





































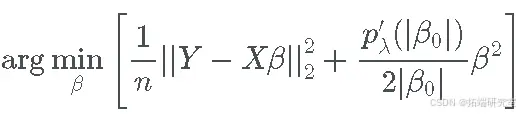





**粗体** _斜体_ [链接](http://example.com) `代码` - 列表 > 引用。你还可以使用@来通知其他用户。